Nuestras maestras, las bacterias
Compartir
Un tren bifronte
Ya lo saben los lectores de estos apuntes: montados en un tren imaginario viajamos rumbo a la célula vegetal en busca de los secretos (y los ya no tan secretos) mecanismos de la vida de las plantas. Un vehículo fantástico de estructura similar a la de una cápsula de simétrica arquitectura, cuyos extremos son dos cabinas de amplios ventanales que resultan frontales o traseras de acuerdo a la dirección que tomamos en cada caso. La que aloje a este cronista será siempre la cabina delantera en compañía de quien nos ayude a describir y entender lo que observamos en cada tramo del viaje, pero no por eso cuentan menos quienes ocupen la que ocasionalmente resulte ser la trasera: viajeros voluntarios que persisten en su generosa asistencia de retaguardia, útil especialmente para evitar ese encandilamiento excesivo que producen algunas revelaciones del conocimiento.
En esta ocasión va con nosotros adelante el Dr. Ramón Enrique, biotecnólogo, investigador del Conicet y parte del equipo de profesionales del Itanoa, dispuesto a introducirnos en los fundamentos de la más nueva y promisoria tecnología de intervención humana en el genoma de las plantas: la edición genómica. En la retaguardia, pacientes, siguen con nosotros Nadia Chalfoun, investigadora del Itanoa y Juan Carlos Díaz Ricci, Director del Instituto Superior de Investigaciones Biológicas (UNT-Conicet), siempre atentos y dispuestos a sostener la amplitud de nuestra mirada.
Un intento que viene de lejos
Desde los orígenes de la agricultura los humanos hemos venido buscando las mejores versiones de las plantas que nos sirven de alimento. El objetivo ha sido siempre la obtención de variedades más rendidoras, más resistentes a vientos, sequías, heladas, enfermedades o plagas, más y mejor adaptables, en definitiva, al contexto productivo en el que pretendemos cultivarlas. Lo hemos hecho primero observando, seleccionando y reproduciendo variantes surgidas naturalmente y al azar; y luego del descubrimiento de las leyes de la herencia y del rol del ADN en la definición de esa identidad esencial de los organismos vivos, induciendo mutaciones mediante radiaciones o estímulos químicos, o con cruzamientos entre variedades de la misma especie con características destacadas para lograr la descendencia que las exprese. Con los avances del conocimiento del ADN esas técnicas han ido perfeccionándose, en busca de la mayor precisión y eficacia posible de esa intervención humana en el genoma de las plantas.
 Impensado desvío
Impensado desvío
Ya hemos visto en imágenes el espectáculo del interior de una célula eucariota, similar al de una ciudad superpoblada de intrincada estructura edilicia, organismos especializados, abigarrada red de interconexiones y un intensísimo tránsito molecular, señales bioquímicas y transporte de agua y residuos metabólicos. Y hemos sido advertidos en nuestras conversaciones previas con Juan Carlos Díaz Ricci y Nadia Chalfoun1 de la importancia que ha adquirido el conocimiento de esos procesos propios del metabolismo celular que tienen lugar en el citoplasma, de su participación protagónica en las respuestas a estímulos ambientales y de lo determinantes que son a la hora de entender la relación de ida y vuelta que hay entre el citoplasma y el ADN, especialmente en la perspectiva de inducir en las especies cultivables mecanismos de autodefensa.
Atraídos ahora por las revelaciones acerca de lo último en materia de intervención humana en el genoma de las plantas, es el núcleo hacia donde nuestra curiosidad elegirá conducirnos, pero no será finalmente el suscripto quien decida el particular destino de nuestra primera parada en esa dirección. Antes, y a sugerencia de nuestro guía esta vez, el Dr. Ramón Enrique, tomaremos un desvío. La invitación es la de observar lo que ocurre a partir del “bacterizaje”de un virus -un bacteriófago- sobre la membrana de una bacteria, a través de la que inyectará su ADN invasor con el propósito de colonizarla y utilizar sus recursos para reproducirse y multiplicarse hasta hacerla estallar (lisis)… si la bacteria invadida se lo permite.
Tal como ocurrió por primera vez hacia finales de la década de 1970, serán las bacterias las que nos marquen el camino de lo que, a partir de aquel momento, comenzaría a conocerse como la tercera revolución verde: la aventura científica de la ingeniería genética. Con el posterior y relativamente reciente desentrañamiento, en 2012,de uno de los varios mecanismos de defensa de las bacterias, denominado sistema CRISPR-Cas2, la manipulación genética de las plantas adquiriría una nueva y alentadora perspectiva.
El gran salto
Ocurrió en 1977. Mary Dell-Chilton, por entonces de 38 años y ya dedicada a la biología molecular, fue quien dio el primer paso. El equipo con el que trabajaba en la Universidad de Washington investigaba el mecanismo por el cual la bacteria Agrobacterium tumefaciens causaba en las plantas que infectaba una mortal tumoración -conocida como agalla de la corona– situada en la zona de transición entre la raíz y el tallo. La evidencia dictaba que lo hacía transfiriendo a la planta un gen maligno que producía la afección, cosa que, en principio, les parecía imposible. Tratando de probar que se trataba de un error, lo que consiguió en cambio Dell-Chilton fue comprobar que, efectivamente, Agrobacterium poseía esa especial habilidad y con ello, que genes bacterianos podían recombinarse con el ADN de los organismos vegetales. A partir de ese descubrimiento, lo que lograron fue sustituir el gen pernicioso por otros que resultaran útiles para inducir en la planta receptora alguna aptitud deseada, aprovechando esa capacidad de la bacteria como vector de la transferencia, sentando de una vez y quizá para siempre las bases de la ingeniería genética y dando origen a lo que hoy conocemos como transgénesis.
Puntería y efectos conducentes
El desafío consiste en obtener los mejores resultados en el menor tiempo posible. Y si bien la respuesta definitiva de lo que logremos mediante la manipulación genética la obtendremos del comportamiento de las nuevas variedades una vez puestas a desarrollarse y a producir en el campo, es mucho lo que, según veremos, podemos avanzar en el laboratorio.
El mejoramiento genético hoy llamado “convencional” consiste, como dijimos, en obtener mediante el cruzamiento de variedades de interés otras descendientes con las características deseadas, puede llevar -como ocurre en especies de genomas complejos como el de la caña de azúcar- hasta 15 años. Y si bien han ido acelerándose los tiempos mediante el uso de marcadores moleculares que permiten identificar en los individuos descendientes la región del genoma en la que se hallen esos genes de interés para saber mejor qué ir cruzando, los tiempos aún son largos y los costos altos. La tentación de intervenir directamente en el ADN vegetal fue -y sigue siendo- comprensiblemente alta, sobre todo teniendo en cuenta los avances de las biotecnologías que la hacen factible.
La cuestión era cómo hacerlo con la mayor precisión posible. Precisión que implica dos aspectos: por un lado, intervenir en el lugar deseado, sin errores, digamos, de “puntería” (“off targets”) que pueden ocurrir en prácticas corrientemente utilizadas, como las del silenciamiento génico o la transgénesis; por el otro, para lograr mediante esa intervención el efecto deseado en el comportamiento de la planta. Este segundo aspecto depende de los progresos del conocimiento del ADN vegetal y de la interacción consecuente con lo que ocurre en el citoplasma. La edición genómica permitiría, en principio, lograr los mismos objetivos pero avanzando en ambos sentidos.
Recombinación genética
La transgénesis, que ha sido hasta aquí de gran utilidad para la producción agrícola al dotar a las plantas de aptitudes que naturalmente no poseían (resistencia a insectos, a sequía, a herbicidas, por ejemplo), consiste, como se sabe, en la incorporación en el genoma vegetal de un gen “extranjero” proveniente de otras especies cultivables, como el maíz, o de otros organismos vivos como insectos o bacterias.
Uno de los métodos utilizados para la incorporación de un gen exógeno en las células destino -técnicas de “delivery”-, además del original utilizando una agrobacteria como vector, es el de la biobalística.
“Nosotros -nos dice Ramón Enrique-, que hacemos transgénesis aquí en el Itanoa, utilizamos la biobalística, un método físico consistente en el transporte de ese material exógeno mediante partículas de oro impulsadas por un chorro de helio a una alta presión. Apuntamos a un tejido desdiferenciado, que llamamos ‘explantos’, obtenido de otro que ya era maduro, pero en el que, a través de repiques en medios de cultivos especializados y con el agregado de hormonas, podemos lograr que esas células, que ya habían decidido ser hojas, vuelvan a ser un tejido totipotencial. Ese es el mejor tejido para incorporar el ADN”.
“Después del ‘bombardeo’ al que hemos sometido esos explantos (necesitaremos contar con miles de esos ‘callitos’), tenemos que ver en cuántos hemos logrado transferir el gen exógeno y en cuáles de esas células impactadas exitosamente el gen de interés prospera hasta probar que los fenotipos resultantes expresan el carácter buscado. Es un proceso de embudo en el que, partiendo de esos miles de callitos, podemos eventualmente llegar a esos poquitos que dan origen al evento deseado. Un proceso complejo, estocástico5, que en definitiva conlleva imprecisiones propias de esa aleatoriedad y de la mortificación biológica a la que habremos sometido a esas células a raíz de ese bombardeo. Algo que a partir del hallazgo del sistema CRISPR-Cas de las bacterias podemos comenzar a superar”.
Con la ayuda del dictado de Ramón Enrique y sirviéndonos -también por su recomendación- de lo que encontramos descripto en una publicación del Instituto de Genómica Innovadora6, intentaremos una descripción lo más sintética posible de los fundamentos de este sistema.
Lecciones bacterianas
Los bacteriófagos son virus pequeños, tenaces adversarios de las bacterias, cuyo único propósito es el de reproducirse a sus expensas. Existe una enorme diversidad de especies de estos virus, presentes también en nuestro organismo, en el que sin producirnos ningún daño cumplen funciones de equilibrio, especialmente en nuestra microbiota intestinal.
Las bacterias han desarrollado distintos mecanismos de defensa ante los intentos de los bacteriófagos por colonizarlas. De estos mecanismos bacterianos, el más sofisticado, “inteligente” y presente en muchas, aunque no en todas, es el que ha dado pista a la hasta aquí más promisoria alternativa para la intervención humana en el genoma de plantas y animales.
El proceso de defensa
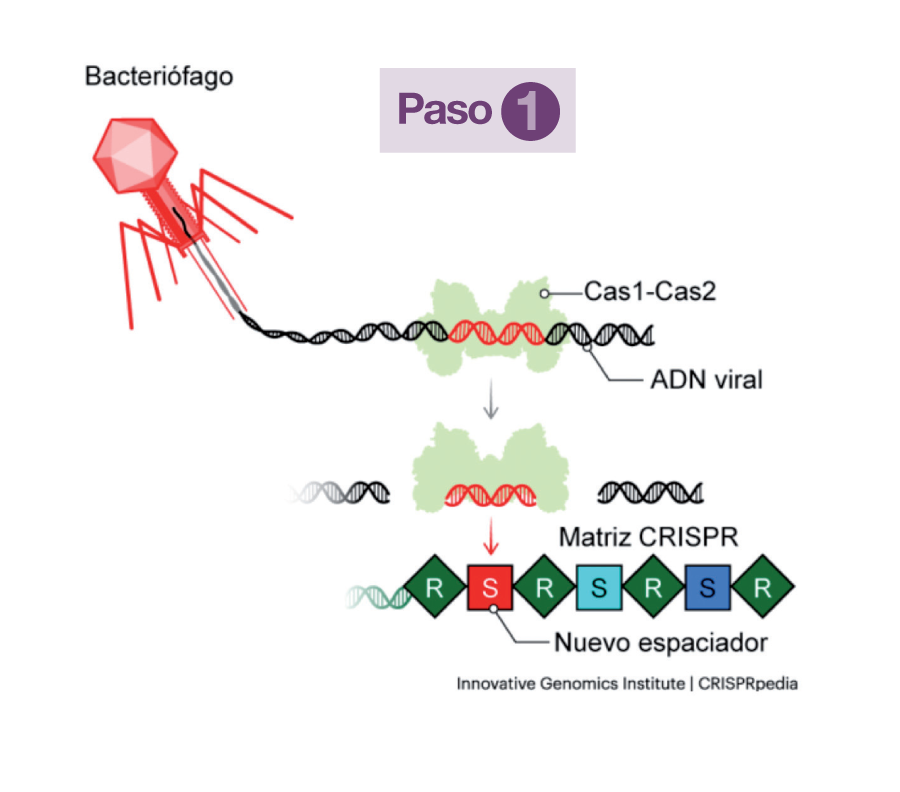
Cada vez que un nuevo bacteriófago inyecta su ADN a través de su membrana, dos proteínas (Cas1 y Cas2) de la bacteria invadida se ocupan de tomar un fragmento del ADN del virus y lo archivan en el suyo, integrándolo en la matriz CRISPR. Ese fragmento del ADN viral se denomina Espaciador (S) y queda enmarcado en dos secuencias repetitivas de ADN bacteriano (R). Esas secuencias Espaciador-Repetición (R-S-R) sirven como memoria defensiva y se transmiten hereditariamente de generación en generación. Tomando como modelo cada espaciador (S), la matriz genera un ARN simple, de una sola hebra, un precursor del crARN, que se unirá a una proteína Cas codificada por una secuencia adyacente a la matriz (Paso 1).
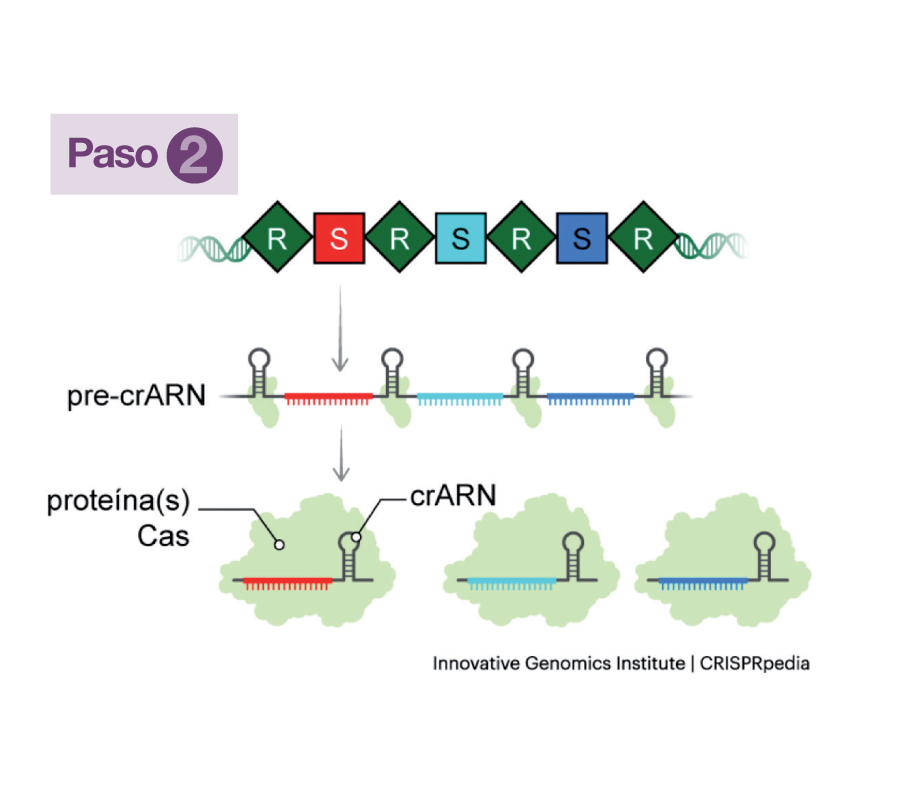
Esa proteína Cas (una nucleasa) unida al precursor, que se convierte así en crARN, conforma con él un complejo de vigilancia en el que el crARN (o ARN guía) es el conductor del “móvil” y la nucleasa, la “policía” (Paso 2).
La misión de estos complejos de vigilancia es circular en el citoplasma bacteriano, detectar la presencia del ADN de un invasor reconocible en el caso de una reinfección y actuar para cortarlo. Una vez hecho esto, otras proteínas de la bacteria se ocupan de destruirlo completamente (Paso 3).
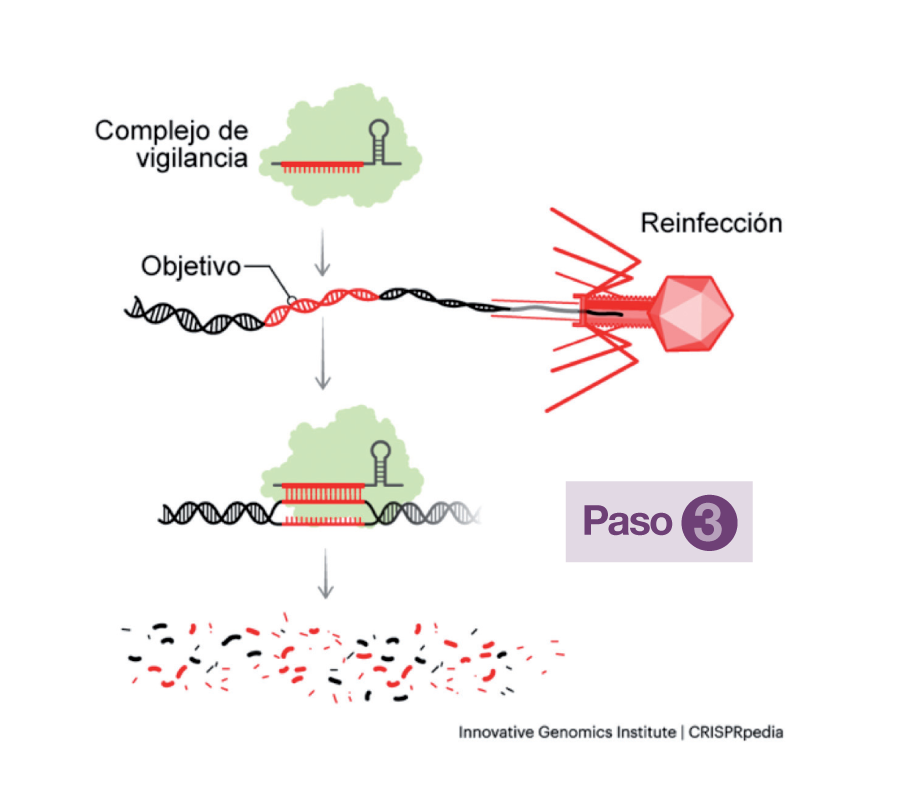 Si estos complejos de defensa no atacan las secuencias guardadas en la matriz CRISPR de la bacteria que las archiva, es porque detectan una marca en el ADN del virus, denominada PAM (Protospacer Adjacent Motifs), presente junto al segmento objetivo del ADN del fago, que la matriz no posee.
Si estos complejos de defensa no atacan las secuencias guardadas en la matriz CRISPR de la bacteria que las archiva, es porque detectan una marca en el ADN del virus, denominada PAM (Protospacer Adjacent Motifs), presente junto al segmento objetivo del ADN del fago, que la matriz no posee.
El viaje continúa
A partir del descubrimiento del sistema CRISPR Cas, la ingeniería genética ha comenzado a desarrollar estos ARN guía, identificar distintos tipos de nucleasas bacterianas (hasta el momento se han identificado 13 diferentes) y a utilizarlas para intervenir con precisión en el genoma de plantas y animales.
¿Cómo y para qué se aplica y cómo recomponen las plantas su genoma intervenido mediante estos cortes de distintas características efectuados ahora con notable puntería? ¿Sirven solo para cortar o también para incorporar otros genes,marcadores o proteínas, útiles para identificar funciones hasta ahora no bien conocidas? ¿Hasta dónde reemplaza esta herramienta otros métodos hasta ahora utilizados? Y más: ¿Cuáles son, a la vista del otro lado de la biología molecular, la de la llamada cuarta revolución verde, los límites, o si se quiere, los interrogantes todavía no resueltos de la ingeniería genética ante la evidencia de la complejidad de los procesos citoplasmáticos? Son preguntas para las que intentaremos encontrar respuestas en una siguiente etapa de este viaje fantástico.
Por ahora, vaya aquel consabido (y en este caso confiable) continuará. Será pues, hasta nuestro próximo encuentro.
1 Ver nuestros primeros dos apuntes en Revista Avance Agroindustrial 43-1 y 43-2 ( https://avance.dev5.thalu.net:82/?articulo=apuntes-de-un-viaje-al-universo-microscopico-de-la-agricultura y https://avance.dev5.thalu.net:82/?articulo=rumbo-a-la-celula )
2 El acrónimo CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats en inglés y Repeticiones Palindrómicas Cortas Agrupadas y Regularmente Espaciadas en español) refiere a una sección del genoma bacteriano llamado matriz CRISPR o matriz de espaciadores repetidos que tiene dos componentes: repeticiones y espaciadores. Esas repeticiones se denominan “palindrómicas” porque contienen un tramo de bases seguido otras complementarias de la misma secuencia, pero en orden inverso, tal como ocurre en el lenguaje, en el que un palíndromo es una frase que puede leerse igual del derecho y del revés, como por ejemplo en la frase luz azul.